반응형
kaggle에 있는 Histopathologic Cancer Detection 대회에서 제공하는 Histopathologic cencer 이미지로 커스텀 데이터셋(custom dataset)을 만들어보도록 하겠습니다.
histopathologic cencer 이미지는 종양이 있는 경우 1, 없는 경우 0 두 가지로 분류되는 이진 분류 문제입니다.
우선, kaggle에서 제공한 데이터 레이블을 확인해보겠습니다. 구글 코랩을 사용했습니다.
import pandas as pd
path2csv = '/content/cookbook/MyDrive/data/train_labels.csv'
labels_df = pd.read_csv(path2csv)
labels_df.head()

id 는 이미지의 이름을 의미합니다.
True와 False 데이터의 비를 확인해보겠습니다.
print(labels_df['label'].value_counts())
labels_df.hist()

몇 가지 sample data를 불러와서 확인해 보겠습니다.
import matplotlib.pylab as plt
from PIL import Image, ImageDraw
import cv2
import numpy as np
import os
# get ids for malignant images
malignantIds = labels_df.loc[labels_df['label']==1]['id'].values
# data is stored here
path2train = '/content/cookbook/MyDrive/data/train/'
# show images in grayscale, if you want color change it to True
color = False
plt.rcParams['figure.figsize'] = (10.0, 10.0)
plt.subplots_adjust(wspace=0, hspace=0)
nrows, ncols = 3, 3
print(malignantIds[:9])
for i,id_ in enumerate(malignantIds[:nrows*ncols]):
full_filenames = os.path.join(path2train, id_ + '.tif')
# load image
img = Image.open(full_filenames)
# draw a 32*32 rectangle
draw = ImageDraw.Draw(img)
draw.rectangle(((32,32),(64,64)),outline='green')
plt.subplot(nrows, ncols, i+1)
if color is True:
plt.imshow(np.array(img))
else:
plt.imshow(np.array(img)[:,:,0],cmap='gray')
plt.axis('off')

histo dataset은 종양이 있는 경우, 32x32 crop된 영역에 종양이 존재합니다.
사각형을 그려서 확인해보았습니다.
이제 custom dataset을 만들어 보겠습니다.
from PIL import Image
import torch
from torch.utils.data import Dataset
# fix torch random seed
torch.manual_seed(0)
class histoCancerDataset(Dataset):
def __init__(self, data_dir, transform, data_type='train'):
# path to images
path2data = os.path.join(data_dir, data_type)
# get a list of images
filenames = os.listdir(path2data)
# get the full path to images
self.full_filenames = [os.path.join(path2data, f) for f in filenames]
# labels are in a csv file named train_labels.csv
csv_filename = data_type + '_labels.csv'
path2csvLabels = os.path.join(data_dir, csv_filename)
labels_df = pd.read_csv(path2csvLabels)
# set data frame index to id
labels_df.set_index('id', inplace=True)
# obtain labels from data frame
self.labels = [labels_df.loc[filename[:-4]].values[0] for filename in filenames]
self.transform = transform
def __len__(self):
# return size of dataset
return len(self.full_filenames)
def __getitem__(self, idx):
# open image, apply transforms and return with label
image = Image.open(self.full_filenames[idx])
image = self.transform(image)
return image, self.labels[idx]
# define a simple transformation that only converts a PIL image into PyTorch tensors
import torchvision.transforms as transforms
data_transformer = transforms.Compose([transforms.ToTensor()])
# define an object of the custom dataset for the train folder
data_dir ='/content/cookbook/MyDrive/data'
histo_dataset = histoCancerDataset(data_dir, data_transformer, 'train')
print(len(histo_dataset))
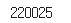
dataset의 길이가 22만개인것을 보아 커스텀 데이터셋이 성공적으로 만들어 졌습니다.
dataset으로부터 이미지를 꺼내서 확인해 보겠습니다.
# load an image
img, label = histo_dataset[9]
print(img.shape, torch.min(img), torch.max(img))
# define a helper function to show an image
def show(img, y, color=False):
# convert tensor to numpy array
npimg = img.numpy()
# Convert to H*W* shape
npimg_tr = np.transpose(npimg, (1,2,0))
if color == False:
npimg_tr = npimg_tr[:,:,0]
plt.imshow(npimg_tr, interpolation='nearest', cmap='gray')
else:
plt.imshow(npimg_tr, interpolation='nearest')
plt.title('title:' + str(y))
show(img, label)

이미지도 성공적으로 불러왔습니다!
이제 train, val dataset으로 나누고 data loader로 감싼뒤에 분석을 할 수 있습니다.
반응형
'Python > PyTorch 공부' 카테고리의 다른 글
| [PyTorch] dataset 분할하기 (2) | 2021.02.28 |
|---|---|
| [PyTorch] convolutional layer 출력 크기 계산하는 함수 만들기 (0) | 2021.02.22 |
| [PyTorch] CNN 신경망 구축하고 MNIST 데이터셋으로 학습하기 (1) | 2021.02.14 |
| [Object Detection] YOLO(v3)를 PyTorch로 바닥부터 구현하기 - Part 5 (0) | 2021.01.31 |
| [Object Detection] YOLO(v3)를 PyTorch로 바닥부터 구현하기 - Part 4 (0) | 2021.01.29 |